پتہ لگانے کی خاصیت
زیادہ تر معاملات میں، پرائمر ڈیزائن کا مقصد پی سی آر کی خصوصیت کو زیادہ سے زیادہ کرنا ہے۔اس کا تعین بہت سے متغیرات کے کم و بیش متوقع اثر سے ہوتا ہے۔ایک اہم متغیر پرائمر کے 3′ آخر میں ترتیب ہے۔
اہم بات یہ ہے کہ مخصوصیت کے لیے بنائے گئے پی سی آر اسیسز وسیع متحرک رینج میں اعلی کارکردگی کو برقرار رکھنے کا زیادہ امکان رکھتے ہیں، کیونکہ پرکھ غیر مخصوص امپلیفیکیشن مصنوعات تیار نہیں کرتی ہے، اس طرح پی سی آر ری ایجنٹس کے ساتھ مقابلہ کرتی ہے یا مرکزی امپلیفیکیشن رد عمل کو روکتی ہے۔
بلاشبہ، بعض صورتوں میں، مخصوصیت سب سے اہم نہیں ہوتی، مثال کے طور پر، جب مقصد قریب سے متعلقہ لیکن مختلف پیتھوجینز کی مقدار درست کرنا ہو، خصوصی ڈیزائن، اصلاح اور تصدیقی معیارات درکار ہوتے ہیں۔
پگھلنے کا وکر ایمپلی کون کی خصوصیت کا اندازہ لگانے کا ایک معیاری طریقہ ہے، کم از کم اس لحاظ سے کہ آیا کسی ایک ہدف کو بڑھانا ہے۔تاہم، اس بات پر زور دینا ضروری ہے کہ پگھلنے والے منحنی خطوط گمراہ کن ہو سکتے ہیں کیونکہ، مثال کے طور پر، وہ سب سے زیادہ پرائمر اور کم ٹیمپلیٹ ارتکاز کے مشترکہ اثرات سے متاثر ہو سکتے ہیں۔
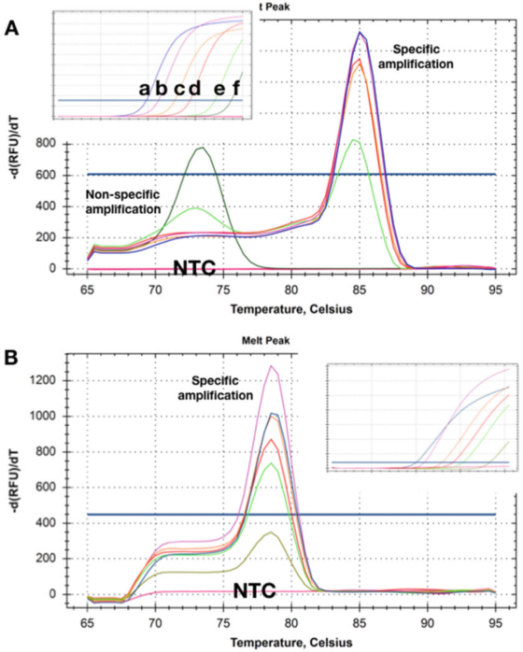
P5 |پگھلنے کا وکر دو ہدف DNAs کی مختلف مقداروں کی دو کھوجوں سے حاصل کردہ Tm شفٹوں کو دکھاتا ہے۔
A. زیادہ ارتکاز (اشتہار) پر، qPCR پیمائش مکمل ہونے کے بعد کوئی واضح پرائمر ڈائمر نہیں ہوتا ہے۔جیسے جیسے ٹیمپلیٹ کا ارتکاز کم ہو کر 50 کاپیاں (e) ہو جاتا ہے، ایک غیر مخصوص پروڈکٹ ظاہر ہونا شروع ہو جاتا ہے اور سب سے کم ارتکاز (f) پر واحد پروڈکٹ بن جاتا ہے۔
B. ٹیسٹ نے تمام ہدف کے ارتکاز پر ایک ہی Tms کو ریکارڈ کیا، اور سب سے کم ارتکاز (5 کاپیاں) پر بھی کوئی واضح پرائمر ڈائمر نہیں تھا۔پتہ لگانے کے ان دو طریقوں کا استعمال کرتے وقت، NTCs میں کسی بھی پرورش کی مصنوعات کا پتہ نہیں چلا۔
P5 نمونوں کے ساتھ حاصل کردہ تحلیل منحنی خطوط کو ظاہر کرتا ہے جس میں ٹیمپلیٹ مختلف ارتکاز میں موجود ہے۔P 5a سے پتہ چلتا ہے کہ دو سب سے کم ارتکاز پر، غیر مخصوص امپلیفیکیشن پروڈکٹس کے ٹی ایم ایس مخصوص ایمپلی کانز سے کم ہیں۔
ظاہر ہے، پتہ لگانے کا یہ طریقہ قابل اعتماد طریقے سے ایسے اہداف کا پتہ لگانے کے لیے استعمال نہیں کیا جا سکتا جو کم ارتکاز میں موجود ہوں۔
دلچسپ بات یہ ہے کہ NTCs، یعنی ایسے نمونے جن میں DNA بالکل بھی نہیں ہے، نے (غیر مخصوص) امپلیفیکیشن پروڈکٹس کو ریکارڈ نہیں کیا، جس سے یہ ظاہر ہوتا ہے کہ بیک گراؤنڈ جینومک DNA غیر مخصوص ایمپلیفیکیشن/پولیمرائزیشن میں حصہ لے سکتا ہے۔
بعض اوقات اس طرح کے پس منظر کے پرائمر اور غیر مخصوص امپلیفیکیشن کا تدارک نہیں کیا جا سکتا، لیکن اکثر یہ ممکن ہوتا ہے کہ پتہ لگانے کا ایسا طریقہ وضع کیا جائے جس میں کسی بھی ٹیمپلیٹ کے ارتکاز اور NTC (P 5b) میں غیر مخصوص ایمپلیفیکیشن نہ ہو۔
یہاں تک کہ 35 کے Cq کے ساتھ ہدف کے ارتکاز کو بڑھانا بھی ایک مخصوص تحلیل وکر پیدا کرے گا۔اسی طرح، NTCs نے غیر مخصوص پروردن کی کوئی علامت نہیں دکھائی۔بعض اوقات، پتہ لگانے کا رویہ ماں کی شراب پر منحصر ہو سکتا ہے، اور بعض بفر کمپوزیشنز میں صرف غیر مخصوص ایمپلیفیکیشن کا پتہ چلا ہے، جو مختلف Mg2+ ارتکاز سے متعلق ہو سکتا ہے۔
پتہ لگانے کا استحکام
Ta کی اصلاح qPCR کا پتہ لگانے کے تجرباتی تصدیق اور اصلاح کے عمل میں ایک مفید قدم ہے۔یہ درجہ حرارت (یا درجہ حرارت کی حد) دکھا کر پرائمر سیٹ کی مضبوطی کا براہ راست اشارہ فراہم کرتا ہے جو NTC کو بڑھاے بغیر سب سے کم Cq پیدا کرتا ہے۔
حساسیت میں دو سے چار گنا فرق زیادہ ایم آر این اے اظہار والے لوگوں کے لیے اہم نہیں ہو سکتا، لیکن تشخیصی ٹیسٹوں کے لیے، اس کا مطلب مثبت اور غلط منفی نتائج کے درمیان فرق ہو سکتا ہے۔
qPCR پرائمر کی Ta خصوصیات بہت مختلف ہو سکتی ہیں۔کچھ ٹیسٹ بہت مضبوط نہیں ہوتے ہیں، اور اگر وہ پرائمر کی بہترین Ta ویلیو کے تحت نہیں کیے جاتے ہیں، تو وہ جلدی سے گر جائیں گے۔
یہ ضروری ہے کیونکہ اس قسم کا پتہ لگانا اکثر حقیقی دنیا میں پریشانی کا باعث ہوتا ہے، اور نمونے کی پاکیزگی، ڈی این اے کا ارتکاز، یا دوسرے ڈی این اے کی موجودگی ممکن نہیں ہے۔
اس کے علاوہ، ٹارگٹ کاپی نمبر وسیع رینج میں مختلف ہو سکتا ہے، اور ری ایجنٹس، پلاسٹک کے برتن، یا آلات ان سے مختلف ہو سکتے ہیں جو ٹیسٹ ترتیب دیتے وقت استعمال کیے جاتے ہیں۔
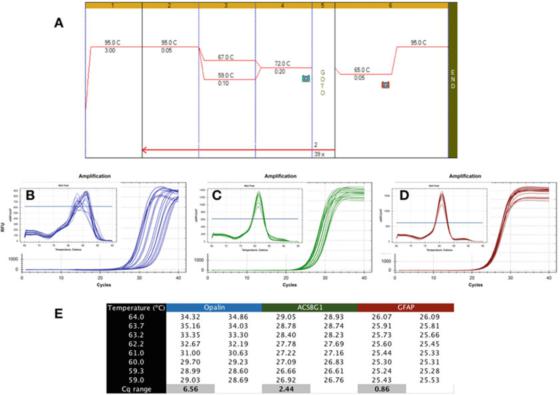
P6|درجہ حرارت کا میلان پی سی آر کا پتہ لگانے کی مختلف مضبوطی کو ظاہر کرتا ہے۔
A. انسانی دماغ کے RNA سے تیار کردہ cDNA پر PCR کرنے کے لیے Bioline's Sensifast SYBR ماسٹر مکس (کیٹلاگ نمبر BIO-98050) استعمال کریں۔
B. Apalene (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGTGGTGATCTCCTAGG) کے ایمپلیفیکیشن میپ اور تحلیل وکر کو ریکارڈ کرنے کے لیے Bio-Rad کا CFX qPCR آلہ استعمال کریں۔
C. ACSBG1 (NM_015162.4, F: CTACACTTCGGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG) کا ایمپلیفیکیشن گراف اور پگھلنے کا وکر۔
D. GFAP کا ایمپلیفیکیشن گراف اور تحلیل وکر (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCTCGTGGATCTTC)۔
E. Cqs مختلف اینیلنگ درجہ حرارت پر ریکارڈ کیے گئے، جو 7C درجہ حرارت کے میلان کے تحت ریکارڈ کیے گئے Cq میں فرق کو ظاہر کرتا ہے۔
P 6 ایک ناپسندیدہ ٹیسٹ کا ایک عام نتیجہ دکھاتا ہے، جہاں qPCR کو 59C اور 67C (P 6a) کے درمیان گریڈینٹ Tas کا استعمال کرتے ہوئے، تین انسانی دماغ کے مخصوص جینز کے لیے پرائمر کا استعمال کرتے ہوئے کیا گیا تھا۔
ایمپلیفیکیشن گراف سے یہ دیکھا جا سکتا ہے کہ Opalin پرائمر مثالی سے بہت دور ہیں کیونکہ ان کی بہترین Ta رینج بہت تنگ ہے (شکل 6b)، یعنی Cqs بڑے پیمانے پر منتشر ہیں، جس کے نتیجے میں Cqs کو ان کے بہترین Cqs کم کے مقابلے میں نمایاں طور پر دیکھا جا سکتا ہے۔
پتہ لگانے کا یہ طریقہ غیر مستحکم ہے اور اس سے سب سے زیادہ پروردن کا باعث بن سکتا ہے۔لہذا، پرائمر کے اس جوڑے کو دوبارہ ڈیزائن کیا جانا چاہئے.اس کے علاوہ، پگھلنے کے منحنی تجزیہ (انسیٹ) سے پتہ چلتا ہے کہ اس پتہ لگانے کے طریقہ کار کی خصوصیت بھی مشکل ہو سکتی ہے، کیونکہ ہر Ta کا پگھلنے کا وکر مختلف ہوتا ہے۔
P 6c میں دکھایا گیا ACSBG1 کا پتہ لگانے کا طریقہ اوپر Opalin کا پتہ لگانے کے طریقہ سے زیادہ مضبوط ہے، لیکن یہ اب بھی مثالی نہیں ہے، اور امکان ہے کہ اسے بہتر کیا جا سکتا ہے۔
تاہم، ہم اس بات پر زور دیتے ہیں کہ مضبوطی اور مخصوصیت کے درمیان کوئی ضروری تعلق نہیں ہے، کیونکہ اس پتہ لگانے کے طریقہ سے پیدا ہونے والا تحلیل وکر تمام Tas (انسیٹ) میں ایک ہی چوٹی کی قدر کو ظاہر کرتا ہے۔
دوسری طرف، مضبوطی کا ٹیسٹ بہت زیادہ روادار ہے، جس سے Tas کی وسیع رینج میں اسی طرح کے Cqs پیدا ہوتے ہیں، جیسا کہ GFAP ٹیسٹ میں P 6d میں دکھایا گیا ہے۔
اسی 8 ڈگری سیلسیس کی حد میں حاصل کردہ Cqs میں فرق 1 سے کم ہے، اور تحلیل وکر (انسیٹ) اس درجہ حرارت کی حد میں پتہ لگانے کی خصوصیات کی تصدیق کرتا ہے۔یہ بات قابل غور ہے کہ حساب شدہ Tas اور اصل Ta کی حد بہت مختلف ہو سکتی ہے۔
محققین کو موثر پرائمر ڈیزائن کرنے میں مدد کرنے کے لیے بہت سی رہنما خطوط تیار کی گئی ہیں، جن میں سے زیادہ تر طویل عرصے سے قائم کردہ اصولوں پر مبنی ہیں اور پرائمرز کے 3′ سرے پر بہت زیادہ توجہ دی گئی ہے۔یہ اکثر تجویز کیا جاتا ہے کہ 3' کے آخر میں ایک G یا C اور دو G یا C بیس (GC کلیمپ) شامل کریں، لیکن آخری 5 بیسز میں سے دو سے زیادہ نہیں۔
عملی طور پر، یہ اصول محققین کی رہنمائی کر سکتے ہیں، لیکن ضروری نہیں کہ وہ تمام حالات میں درست ہوں۔
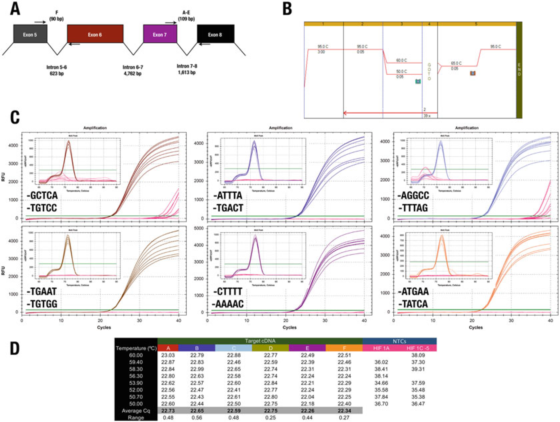
P7 |پرائمر کے 3′سرے کا خاصیت یا کارکردگی پر بہت کم اثر پڑتا ہے۔
A. انسانی HIF-1α (NM_181054.2) جین کے لیے پرائمر کی پوزیشن۔
B. چھ ٹیسٹ اشیاء کو بڑھانے کے لیے Agilent Brilliant III SYBR گرین مادر شراب (Cat. No. 600882) استعمال کریں۔
C. Bio-Rad کے CFX qPCR آلے اور 3′end پرائمر کے ذریعے ریکارڈ کردہ ایمپلیفیکیشن گراف اور پگھلنے کا وکر۔NTCs کو سرخ رنگ میں دکھایا گیا ہے۔
D. ہر ٹیسٹ آئٹم کا Cqs ریکارڈ
مثال کے طور پر، P 7 کا نتیجہ 3′end کے اصول سے متصادم ہے۔تمام ڈیزائن بنیادی طور پر ایک جیسے نتائج پیدا کرتے ہیں، صرف دو پرائمر امتزاج کے ساتھ NTC میں غیر مخصوص امپلیفیکیشن کا باعث بنتے ہیں۔
تاہم، ہم GC کلپ کے اثر کی حمایت نہیں کر سکتے، کیونکہ اس صورت میں، A یا T کو زیادہ سے زیادہ 30 اڈوں کے طور پر استعمال کرنے سے مخصوصیت کم نہیں ہوتی ہے۔
ٹیسٹ C، جہاں F پرائمر GGCC میں ختم ہوتا ہے، نے NTCs میں Cqs کو ریکارڈ کیا، جس سے یہ ظاہر ہوتا ہے کہ کوئی بھی 30 کے آخر میں ان ترتیبوں سے بچنا چاہتا ہے۔ہم اس بات پر زور دیتے ہیں کہ پرائمر جوڑے کے بہترین 3′آخر کی ترتیب کا تعین کرنے کا واحد طریقہ کچھ امیدواروں کے پرائمر کا تجرباتی طور پر جائزہ لینا ہے۔
امپلیفیکیشن کی کارکردگی
اہم بات یہ ہے کہ، اگرچہ غیر مخصوص پی سی آر کا پتہ لگانا کبھی بھی مخصوص نہیں ہو سکتا، لیکن انزائم، مادر شراب، اضافی اشیاء، اور سائیکلنگ کے حالات کو تبدیل کر کے کئی مختلف طریقوں سے ایمپلیفیکیشن کی کارکردگی کو ایڈجسٹ اور زیادہ سے زیادہ کیا جا سکتا ہے۔
پی سی آر کا پتہ لگانے کی کارکردگی کو جانچنے کے لیے، ہدف نیوکلک ایسڈ سے 10 یا 5 گنا سیریل ڈائلیشن کا استعمال کرنا بہتر ہے، یعنی "معیاری منحنی طریقہ"۔
اگر پی سی آر ایمپلیکونز یا مصنوعی ڈی این اے اہداف ایک معیاری منحنی خطوط پیدا کرنے کے لیے استعمال کیے جاتے ہیں، تو ان اہداف کے سیریل ڈائیوشنز کو بیک گراؤنڈ ڈی این اے کی مستقل مقدار (جیسے جینومک ڈی این اے) کے ساتھ ملایا جانا چاہیے۔
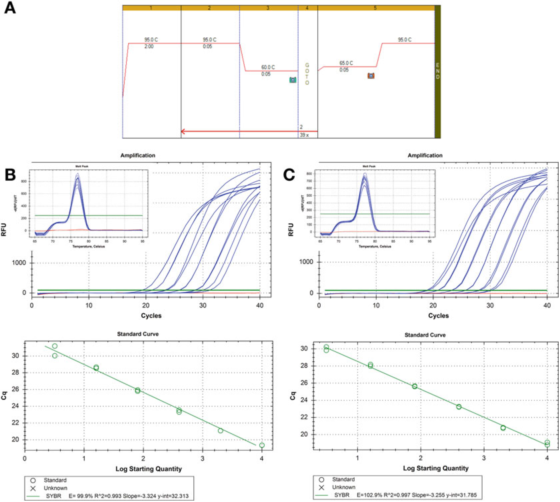
P8 |پی سی آر کی کارکردگی کو جانچنے کے لیے کمزوری وکر۔
A. HIF-1: F: AAGAACTTTTAGGCCGCTCA اور R: TGTCCTGTGTGGTGACTTGTCC اور Agilent's Brilliant III SYBR گرین ماسٹر مکس (کیٹلاگ نمبر 600882) PCR اور پگھلنے والے منحنی حالات کے لیے پرائمر استعمال کریں۔
B. 100 ng RNA کو الٹا نقل کیا گیا تھا، 2 بار پتلا کیا گیا تھا، اور سیریلی طور پر پتلا کیے گئے cDNA نمونوں کو 5 بار 1 ng انسانی جینومک DNA میں پتلا کیا گیا تھا۔پگھلنے کا وکر انسیٹ میں دکھایا گیا ہے۔
C. دوسرے سی ڈی این اے نمونے کے لیے RT ردعمل، کمزوری، اور سیریل ڈیلیوشن کو دہرایا گیا، اور نتائج ایک جیسے تھے۔
P 8 دو معیاری منحنی خطوط دکھاتا ہے، دو مختلف cDNA نمونوں پر ایک ہی پتہ لگانے کا طریقہ استعمال کرتے ہوئے، نتیجہ ایک ہی کارکردگی ہے، تقریباً 100%، اور R2 قدر بھی ملتی جلتی ہے، یعنی تجرباتی ڈیٹا اور ریگریشن لائن کے درمیان فٹ ہونے کی ڈگری یا ڈیٹا کی لکیریت کی ڈگری۔
دو معیاری منحنی خطوط موازنہ ہیں، لیکن بالکل ایک جیسے نہیں ہیں۔اگر مقصد ہدف کی درست مقدار کا تعین کرنا ہے، تو یہ یاد رکھنا چاہیے کہ غیر یقینی صورتحال کی وضاحت کیے بغیر کاپی نمبر کیلکولیشن فراہم کرنا ناقابل قبول ہے۔
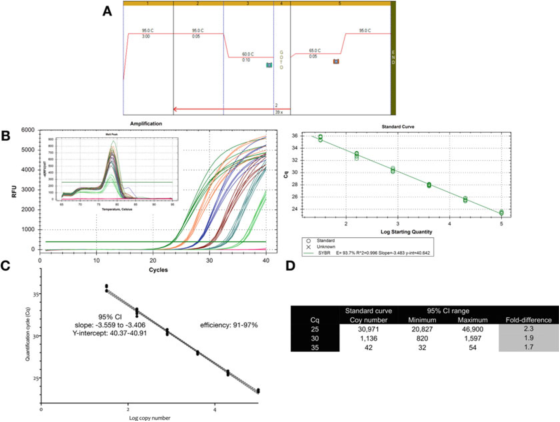
P9 |معیاری وکر کا استعمال کرتے ہوئے مقدار کے تعین سے وابستہ پیمائش کی غیر یقینی صورتحال.
A. PCR اور پگھلنے والے منحنی خطوط کو انجام دینے کے لیے GAPDH (NM_002046) کے لیے پرائمر استعمال کریں۔F: ACAGTTGCCATGTAGACC اور R: TAACTGGTTGAGCACAGG اور Bioline's Sensifast SYBR ماسٹر مکس (کیٹلاگ نمبر BIO-98050)۔
B. ایمپلیفیکیشن چارٹ، پگھلنے کا منحنی خطوط اور معیاری وکر جو Bio-Rad کے CFX qPCR آلہ کے ساتھ ریکارڈ کیا گیا ہے۔
C. معیاری وکر گراف اور 95% اعتماد کا وقفہ (CI)۔
D. کمزوری وکر سے اخذ کردہ تین Cq اقدار کا کاپی نمبر اور 95% اعتماد کا وقفہ۔
P 9 ظاہر کرتا ہے کہ ایک بہتر ٹیسٹ کے لیے، ایک معیاری وکر کی موروثی تغیر تقریباً 2 گنا ہے (95% اعتماد کا وقفہ، کم از کم سے زیادہ سے زیادہ)، جو کہ سب سے چھوٹی تغیر ہو سکتی ہے جس کی توقع کی جا سکتی ہے۔
متعلقہ مصنوعات:
اینیمل ٹشو ڈائریکٹ پی سی آر کٹ
پوسٹ ٹائم: ستمبر 30-2021