جائزہ
ٹرانسجینک پودوں کی تیزی سے شناخت
متن/ٹونگ یوچینگ
تجرباتی آپریشن/ہان ینگ
ایڈیٹر/ وین یوجن
الفاظ/1600+
تجویز کردہ پڑھنے کا وقت/8-10 منٹ
ٹرانسجینک پودوں کی تیزی سے شناخت
لیبارٹری میں ایک نئے آنے والے کے طور پر، کم تبدیلی کی شرح کے ساتھ پودوں کے ایک گروپ سے مثبت پودوں کی اسکریننگ کرنا اچھا کام نہیں ہے۔سب سے پہلے، ڈی این اے کو ایک ایک کرکے بڑی تعداد میں نمونوں سے نکالنا ہوگا، اور پھر پی سی آر کے ذریعے غیر ملکی جینز کا پتہ لگایا جائے گا۔تاہم، نتائج اکثر خالی ہوتے ہیں اور کبھی کبھار کچھ آئٹمز کے ساتھ بینڈ ہوتے ہیں، لیکن اس بات کا تعین کرنا ناممکن ہے کہ آیا کھوئی ہوئی کھوجیں ہیں یا غلط کھوج۔.کیا ایسے تجرباتی عمل اور نتائج کا سامنا کرنا بہت بے بس ہے؟پریشان نہ ہوں، بھائی آپ کو سکھاتا ہے کہ ٹرانسجینک مثبت پودوں کو آسانی سے اور درست طریقے سے کیسے نکالا جائے۔
مرحلہ نمبر 1: ڈیزائن کا پتہ لگانے کے پرائمر
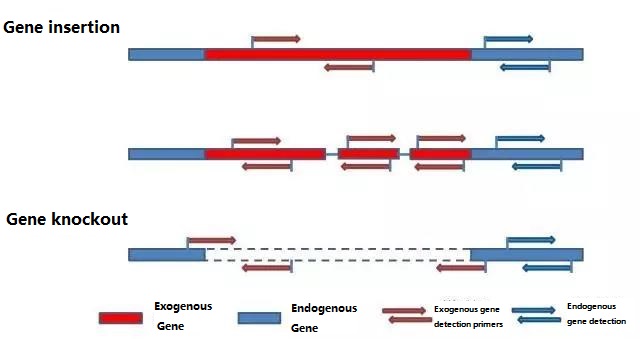
ٹیسٹ کیے جانے والے نمونے کے مطابق پتہ لگانے کے لیے endogenous جین اور exogenous جین کا تعین کریں، اور پرائمر ڈیزائن کے لیے جین میں نمائندہ 100-500bp ترتیب منتخب کریں۔اچھے پرائمر پتہ لگانے کے نتائج کی درستگی کو یقینی بنا سکتے ہیں اور پتہ لگانے کے وقت کو کم کر سکتے ہیں (عام طور پر استعمال ہونے والے پتہ لگانے والے پرائمر کے لیے اپینڈکس دیکھیں)۔
نوٹ:
نئے ڈیزائن کردہ پرائمر کو رد عمل کے حالات کو بہتر بنانے اور بڑے پیمانے پر پتہ لگانے سے پہلے پتہ لگانے کی درستگی، درستگی اور پتہ لگانے کی حد کی تصدیق کرنے کی ضرورت ہے۔
مرحلہ 2:تجرباتی پروٹوکول تیار کریں۔

مثبت کنٹرول: پی سی آر ری ایکشن سسٹم اور حالات نارمل ہیں یا نہیں اس کا تعین کرنے کے لیے ٹارگٹ فریگمنٹ پر مشتمل پیوریفائیڈ ڈی این اے کو بطور ٹیمپلیٹ استعمال کریں۔
منفی/خالی کنٹرول: ڈی این اے ٹیمپلیٹ یا ڈی ڈی ایچ استعمال کریں۔2O جس میں ہدف کا ٹکڑا بطور ٹیمپلیٹ شامل نہیں ہے تاکہ یہ معلوم کیا جا سکے کہ آیا PCR سسٹم میں آلودگی کا کوئی ذریعہ ہے۔
اندرونی حوالہ کنٹرول: نمونے کے اینڈوجینس جین کے پرائمر/تحقیقات کے امتزاج کا استعمال کریں تاکہ جانچ کی جاسکے کہ آیا پی سی آر کے ذریعہ ٹیمپلیٹ کا پتہ لگایا جاسکتا ہے۔
نوٹ:
تجرباتی نتائج کی درستگی کا اندازہ لگانے کے لیے ہر ٹیسٹ کے لیے مثبت، منفی/خالی کنٹرولز اور اندرونی کنٹرول کے کنٹرولز کو سیٹ کیا جانا چاہیے۔
مرحلہ 3: تجرباتی تیاری

استعمال کرنے سے پہلے، دیکھیں کہ آیا حل یکساں طور پر ملا ہوا ہے۔اگر بارش پائی جاتی ہے، تو اسے استعمال کرنے سے پہلے ہدایات کے مطابق تحلیل اور مکس کرنے کی ضرورت ہے۔آئن کی غیر مساوی تقسیم سے بچنے کے لیے 2×PCR مکس کو استعمال کرنے سے پہلے ایک مائیکرو پیپیٹ کے ساتھ بار بار پائپیٹ اور مکس کرنے کی ضرورت ہے۔
نوٹ:
ہدایات کو نکالیں اور انہیں غور سے پڑھیں، اور تجربات سے پہلے ہدایات کے مطابق سختی سے تیاری کریں۔
مرحلہ 4: پی سی آر ری ایکشن سسٹم تیار کریں۔
تجرباتی پروٹوکول کے مطابق، پرائمر مکس کریں، H2O، 2×PCR مکس کریں، سینٹری فیوج کریں اور انہیں ہر ری ایکشن ٹیوب میں تقسیم کریں۔
نوٹ:
بڑے پیمانے پر یا طویل مدتی جانچ کے لیے، UNG انزائم پر مشتمل PCR رد عمل کا نظام استعمال کرنے کی سفارش کی جاتی ہے، جو PCR مصنوعات کی وجہ سے ہونے والی ایروسول آلودگی سے مؤثر طریقے سے بچ سکتا ہے۔
مرحلہ 5: ردعمل ٹیمپلیٹ شامل کریں۔

ڈائریکٹ پی سی آر ٹکنالوجی کا استعمال کرتے ہوئے ، نیوکلک ایسڈ صاف کرنے کے تکلیف دہ عمل کی ضرورت نہیں ہے۔نمونہ ٹیمپلیٹ 10 منٹ کے اندر تیار کیا جا سکتا ہے اور متعلقہ PCR ردعمل کے نظام میں شامل کیا جا سکتا ہے۔
نوٹ:
Lysis طریقہ کار کا پتہ لگانے کا بہتر اثر ہوتا ہے، اور حاصل کردہ پروڈکٹ کو ایک سے زیادہ پتہ لگانے کے رد عمل کے لیے استعمال کیا جا سکتا ہے۔
5.1: پتوں کا براہ راست پی سی آر
دستی میں تصویر کے سائز کے مطابق، 2-3 ملی میٹر قطر کے ساتھ پتی کے ٹشو کو کاٹ کر پی سی آر ری ایکشن سسٹم میں رکھیں۔
نوٹ: اس بات کو یقینی بنائیں کہ پتی کے ٹکڑے مکمل طور پر پی سی آر کے رد عمل کے محلول میں ڈوب گئے ہیں، اور ضرورت سے زیادہ پتی کے ٹشو شامل نہ کریں۔
5.2: لیف لیسز کا طریقہ
پتی کے ٹشو کو 5-7 ملی میٹر قطر کے ساتھ کاٹیں اور اسے سینٹری فیوج ٹیوب میں رکھیں۔اگر آپ پختہ پتوں کا انتخاب کرتے ہیں، تو براہ کرم پتے کی مرکزی رگ کے ٹشوز کو استعمال کرنے سے گریز کریں۔Pipette 50ul Buffer P1 lysate کو ایک سینٹری فیوج ٹیوب میں اس بات کو یقینی بنانے کے لیے کہ lysate پتی کے ٹشو کو مکمل طور پر غرق کر سکتا ہے، اسے تھرمل سائیکلر یا دھاتی غسل میں رکھ سکتا ہے، اور 5-10 منٹ کے لیے 95 ° C پر لیزیٹ کر سکتا ہے۔


50ul Buffer P2 نیوٹرلائزیشن سلوشن شامل کریں اور اچھی طرح مکس کریں۔نتیجے میں lysate ایک ٹیمپلیٹ کے طور پر استعمال کیا جا سکتا ہے اور PCR ردعمل کے نظام میں شامل کیا جا سکتا ہے.
نوٹ: ٹیمپلیٹ کی مقدار PCR سسٹم کے 5-10% کے درمیان ہونی چاہیے، اور 20% سے زیادہ نہیں ہونی چاہیے (مثال کے طور پر، 20μl PCR سسٹم میں، 1-2μl lysis بفر شامل کریں، 4μl سے زیادہ نہیں)۔
مرحلہ 6: پی سی آر رد عمل

پی سی آر ری ایکشن ٹیوب کو سینٹرفیوگ کرنے کے بعد، انہیں پی سی آر آلے میں ایمپلیفیکیشن کے لیے رکھیں۔
نوٹ:
رد عمل ایمپلیفیکیشن کے لیے نان پیوریفائیڈ ٹیمپلیٹ کا استعمال کرتا ہے، اس لیے ایمپلیفیکیشن سائیکلوں کی تعداد پیوریفائیڈ ڈی این اے ٹیمپلیٹ استعمال کرنے کے مقابلے میں 5-10 زیادہ ہے۔
مرحلہ 7: الیکٹروفورسس کا پتہ لگانا اور نتائج کا تجزیہ

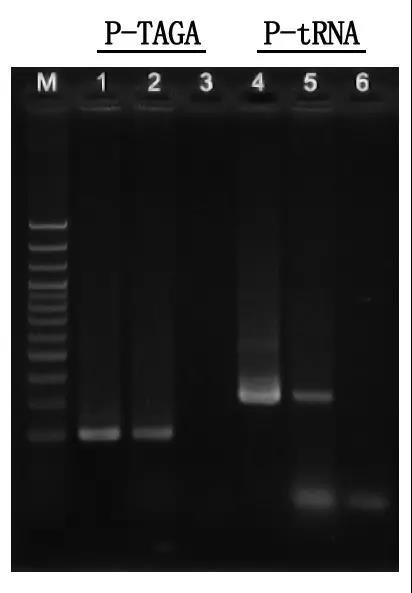
M:100bp DNA سیڑھی
1\4: Purified DNA طریقہ
2\5: براہ راست PCR طریقہ
3\6: خالی کنٹرول
کوالٹی کنٹرول:
تجربے میں سیٹ کیے گئے مختلف کنٹرولز کے ٹیسٹ کے نتائج کو درج ذیل شرائط کو پورا کرنا چاہیے۔دوسری صورت میں، مسئلہ کی وجہ کا تجزیہ کیا جانا چاہئے، اور مسئلہ کو ختم کرنے کے بعد دوبارہ ٹیسٹ کیا جانا چاہئے.
ٹیبل 1. مختلف کنٹرول گروپس کے عام ٹیسٹ کے نتائج

*جب پلاسمڈ کو مثبت کنٹرول کے طور پر استعمال کیا جاتا ہے، تو اینڈوجینس جین ٹیسٹ کا نتیجہ منفی ہو سکتا ہے۔
نتیجہ کا فیصلہ:
A. نمونے کے اینڈوجینس جین کے ٹیسٹ کا نتیجہ منفی ہے، جس سے ظاہر ہوتا ہے کہ عام پی سی آر کا پتہ لگانے کے لیے موزوں ڈی این اے نمونے سے نہیں نکالا جا سکتا یا نکالے گئے ڈی این اے میں پی سی آر ری ایکشن روکنے والے شامل ہیں، اور ڈی این اے کو دوبارہ نکالا جانا چاہیے۔
B. نمونے کے اینڈوجینس جین کے ٹیسٹ کا نتیجہ مثبت ہے، اور خارجی جین کے ٹیسٹ کا نتیجہ منفی ہے، جس سے ظاہر ہوتا ہے کہ نمونے سے عام PCR کا پتہ لگانے کے لیے موزوں DNA نکالا گیا ہے، اور یہ اندازہ لگایا جا سکتا ہے کہ نمونے میں XXX جین کا پتہ نہیں چلا ہے۔
C. نمونے کے اینڈوجینس جین کا ٹیسٹ نتیجہ مثبت ہے، اور exogenous جین کے ٹیسٹ کا نتیجہ مثبت ہے، جس سے ظاہر ہوتا ہے کہ عام PCR کا پتہ لگانے کے لیے موزوں DNA نمونے سے نکالا گیا ہے، اور نمونے کے DNA میں XXX جین موجود ہے۔تصدیقی تجربات مزید کئے جا سکتے ہیں۔
مرحلہ 8: ڈیزائن کا پتہ لگانے والے پرائمر

تجربے کے بعد، ماحولیاتی آلودگی کو روکنے کے لیے تجرباتی علاقے کو صاف کرنے کے لیے 2% سوڈیم ہائپوکلورائٹ محلول اور 70% ایتھنول محلول استعمال کریں۔
اپینڈکس
جدول 2۔ جینیاتی طور پر تبدیل شدہ پودوں کے پی سی آر کا پتہ لگانے کے لیے عام طور پر استعمال ہونے والے پرائمر
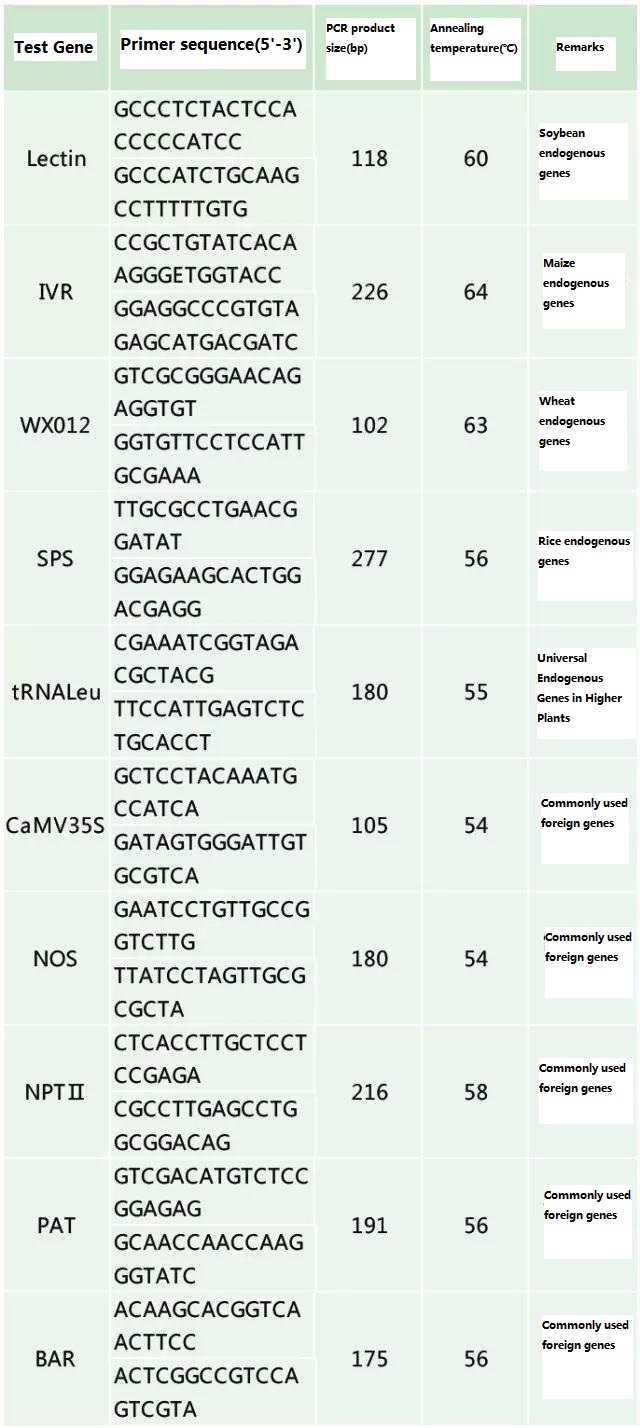
حوالہ دستاویز:
SN/T 1202-2010، خوراک میں جینیاتی طور پر تبدیل شدہ پودوں کے اجزاء کے لیے کوالیٹیٹو PCR کا پتہ لگانے کا طریقہ۔
وزارت زراعت کا اعلان 1485-5-2010، جینیاتی طور پر تبدیل شدہ پودوں اور ان کی مصنوعات کے اجزاء کی جانچ - چاول M12 اور اس کے مشتقات۔
پوسٹ ٹائم: جون 09-2021








